RNA-Seq
RNA-seq (secvențierea ARN, în engleză RNA sequencing), denumită și secvențierea shotgun a unui întreg transcriptom [1], este o tehnologie care utilizează secvențierea de nouă generație pentru a identifica și cuantifica ARN-ul dintr-un genom la un moment dat. [2]
Introducere[modificare | modificare sursă]
Transcriptomul unei celule este dinamic, se schimbă continuu. Tehnologiile de secvențiere de nouă generație (engleză next generation sequencing) au permis citirea ADN-ului dintr-o celulă, bază cu bază. Aceste tehnologii permit și citirea ARN-ului dintr-o celulă, fapt care facilitează înțelegerea splicingului alternativ al genelor, înțelegerea modificărilor post-transcripție, fuziunea genelor, polimorfismul uninucleotidic (SNP) și schimbările în exprimarea genetică.[3] În plus față de ARN-ul mesager, tehnologia RNA-Seq identifică și alte tipuri de ARN, cum ar fi microARN (engleză miRNA), ARN de transfer (engleză tRNA) și ARN ribozomal (engleză rRNA). [4]
Tehnologia RNA-Seq este utilă pentru determinarea marginilor exon/intron și pentru verificarea capetelor genelor 5’ și 3’.
Cercetarea care se face cu ajutorul tehnologiei RNA-Seq include observarea schimbărilor care au loc într-o celulă în timpul infecțiilor [5] și schimbările din exprimarea genetică în studiul cancerului.[6]
Înainte de modernizarea tehnologiile de secvențiere de nouă generație, studierea exprimării genetice se făcea cu tehnologia microarray.
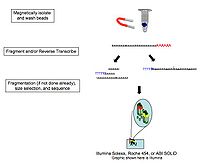
Metodologie[modificare | modificare sursă]

Referințe[modificare | modificare sursă]
- ^ Ryan D. Morin, Matthew Bainbridge, Anthony Fejes, Martin Hirst, Martin Krzywinski, Trevor J. Pugh, Helen McDonald, Richard Varhol, Steven J.M. Jones, and Marco A. Marra. (). „Profiling the HeLa S3 transcriptome using randomly primed cDNA and massively parallel short-read sequencing”. BioTechniques. 45 (1): 81–94. doi:10.2144/000112900. PMID 18611170. Arhivat din original la . Accesat în .
- ^ Chu Y, Corey DR (august 2012). „RNA sequencing: platform selection, experimental design, and data interpretation”. Nucleic Acid Ther. 22 (4): 271–4. doi:10.1089/nat.2012.0367. PMC 3426205
 . PMID 22830413.
. PMID 22830413.
- ^ Maher CA, Kumar-Sinha C, Cao X, et al. (martie 2009). „Transcriptome sequencing to detect gene fusions in cancer”. Nature. 458 (7234): 97–101. doi:10.1038/nature07638. PMC 2725402
 . PMID 19136943.
. PMID 19136943.
- ^ Ingolia NT, Brar GA, Rouskin S, McGeachy AM, Weissman JS (august 2012). „The ribosome profiling strategy for monitoring translation in vivo by deep sequencing of ribosome-protected mRNA fragments”. Nat Protoc. 7 (8): 1534–50. doi:10.1038/nprot.2012.086. PMC 3535016
 . PMID 22836135.
. PMID 22836135.
- ^ Qian F, Chung L, Zheng W, et al. (). „Identification of Genes Critical for Resistance to Infection by West Nile Virus Using RNA-Seq Analysis”. Viruses. 5 (7): 1664–81. doi:10.3390/v5071664. PMID 23881275.
- ^ Beane J, Vick J, Schembri F, et al. (iunie 2011). „Characterizing the impact of smoking and lung cancer on the airway transcriptome using RNA-Seq”. Cancer Prev Res (Phila). 4 (6): 803–17. doi:10.1158/1940-6207.CAPR-11-0212. PMC 3694393
 . PMID 21636547.
. PMID 21636547.
